MicroRNA ketma-ketligi - MicroRNA sequencing - Wikipedia
MicroRNA ketma-ketligi (miRNA-seq), turi RNK-sek, foydalanish keyingi avlod ketma-ketligi yoki katta parallel yuqori o'tkazuvchanlik DNKning ketma-ketligi ketma-ketlikka mikroRNKlar, shuningdek, miRNAlar deb ataladi. miRNA-seq boshqa RNK-seq shakllaridan farq qiladi, chunki kirish materiali ko'pincha kichik RNKlar uchun boyitiladi. miRNA-seq tadqiqotchilarga to'qimalarga xos ekspression shakllarini, kasallik assotsiatsiyasini va miRNAlarning izoformalarini o'rganishga va ilgari xarakterlanmagan miRNKlarni kashf qilishga imkon beradi. Kabi tartibga solinmagan miRNKlarning rolini o'ynaydigan dalillar saraton[1] miRNA-seq ni kelajakda diagnostika va prognozlash uchun muhim vosita bo'lishi uchun joylashtirdi, chunki xarajatlar kamayib bormoqda.[2] Boshqa miRNA profillash texnologiyalari singari, miRNA-Seq ham afzalliklarga ega (ketma-ketlik mustaqilligi, qamrovi) va kamchiliklari (yuqori narx, infratuzilma talablari, ishlash muddati va potentsiali asarlar ).[3]
Kirish
MikroRNKlar (miRNAlar) - uzunligi 21-25 nukleotid bo'lgan, transkriptning parchalanishi, inhibisyoni orqali oqsil ekspluatatsiyasini modulyatsiya qiladigan kichik ribonuklein kislotalarining oilasi. tarjima yoki transkriptlarni sekvestrlash.[4][5][6] Birinchi kashf etilgan miRNA, lin-4, genetikada topilgan mutagenez nematodaning embriondan keyingi rivojlanishini boshqaruvchi molekulyar elementlarni aniqlash uchun ekran Caenorhabditis elegans.[7] The lin-4 gen 22-nukleotidli RNKni 3'-tarjima qilinmagan mintaqasida konservatsiyalangan bog'lanish joylari bilan kodlagan. lin-14 mRNA stenogramma[8] va regulyatsiya qilingan LIN-14 oqsil ekspressioni.[9] Hozirda miRNKlar ko'plab rivojlanish va biologik jarayonlarni, shu jumladan tartibga solishda ishtirok etadi deb o'ylashadi gemopoez (miR-181 yilda Muskul mushak[10]), lipid metabolizmi (miR-14 yilda Drosophila melanogaster[11]) va neyronlarning rivojlanishi (lsy-6 yilda Caenorhabditis elegans[12]).[6] Ushbu kashfiyotlar miRNA-seq kabi miRNKlarni aniqlash va tavsiflashga qodir bo'lgan texnikani ishlab chiqishni talab qildi.
Tarix
Fursatdan foydalanish uchun MicroRNA sekvensiyasi (miRNA-seq) ishlab chiqilgan keyingi avlod ketma-ketligi yoki katta darajada parallel yuqori o'tkazuvchanlik ketma-ketligi yangi miRNAlarni va ularning ekspression rejimlarini berilgan namunada topish uchun texnologiyalar. miRNA sekvensiyasi o'zi uchun yangi g'oya emas, ketma-ketlikning dastlabki usullari qo'llaniladi Sanger ketma-ketligi usullari. Tartibga tayyorlash kutubxonalarni yaratishni o'z ichiga oladi klonlash ning DNK ustun bilan tanlangan 21-25 bp o'lchamdagi endogen kichik RNKlardan teskari transkripsiya qilingan gel elektroforezi.[13] Biroq, bu usul vaqt va resurslar nuqtai nazaridan to'liqdir, chunki har bir klon alohida kuchaytirilishi va ketma-ketlikka tayyorlanishi kerak. Ushbu usul ham beixtiyor yuqori darajada ifoda etilgan miRNKlarga yordam beradi.[6] Keyingi avlod ketma-ketligi ketma-ketlikda zarur bo'lgan gibridizatsiya zondlariga ehtiyojni yo'q qiladi DNK mikroarray tahlil qilish, shuningdek Sanger ketma-ketligi usulida talab qilinadigan mashaqqatli klonlash usullari. Bundan tashqari, miRNA-SEQ usulidagi yangi avlod ketma-ketlik platformalari kichik RNKlarning katta hovuzlarini bitta ketma-ketlikda bajarilishini osonlashtiradi.[14]
miRNA-seq turli ketma-ketlik platformalari yordamida amalga oshirilishi mumkin. Kichiklarning birinchi tahlili RNKlar miRNA-seq usullari yordamida model zavodidan taxminan 1,4 million kichik RNK tekshirildi Arabidopsis talianasi foydalanish Lynx Therapeutics-ning ommaviy ravishda parallel ravishda imzolanishi (MPSS) ketma-ketlik platformasi. Ushbu tadqiqot kichik RNKlarni o'rganish uchun yangi, yuqori o'tkazuvchanlik ketma-ketligi texnologiyalarining potentsialini namoyish etdi va genomlar kichik RNKlarning boy manbalari sifatida o'simliklar bilan ko'p sonli kichik RNKlarni hosil qilishini ko'rsatdi.[15] Keyinchalik tadqiqotlar boshqa ketma-ketlik texnologiyalaridan foydalanilgan, masalan C. elegans 18 yangi miRNA genini, shuningdek yangi nematodli kichik RNK sinfini aniqladi 21U-RNK.[16] Odamning bachadon bo'yni o'smalari va normal to'qimalarining kichik RNK profillarini taqqoslaydigan yana bir tadqiqotda ishlatilgan Illumina (kompaniya) Genom analizatori insonning 64 ta yangi miRNA genini hamda 67 ta differentsial ekspresiya qilingan miRNA ni aniqlash uchun.[17] Amaliy biosistemalar SOLiD sekvensiya platformasi, shuningdek, odamning ko'krak bezi saratonini aniqlashda miRNAlarning prognostik qiymatini o'rganish uchun ishlatilgan.[18]
Usullari
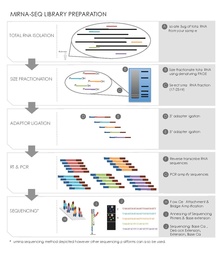
Kichik RNK preparati
Ketma-ket kutubxonani qurish yuqori ishlaydigan ketma-ketlik platformasiga qarab turli xil to'plamlar yordamida amalga oshirilishi mumkin. Shu bilan birga, kichik RNK sekvensiyasini tayyorlash uchun bir nechta umumiy qadamlar mavjud.[19][20]
RNKning umumiy izolatsiyasi
Berilgan namunada barcha RNK izotiyosiyanat / fenol / xloroform (GITC / fenol) usuli yoki Trizol (masalan, tijorat mahsuloti) yordamida ajratib olinadi va ajratib olinadi.Invitrogen ) reaktiv. Jelni tozalash va hajmini tanlash uchun odatda boshlang'ich miqdori 50-100 mkg bo'lgan umumiy RNK, 1 g to'qima odatda 1 mg umumiy RNK hosil qiladi.[20] RNK sifatini boshqarish ham o'lchanadi, masalan, Caliper LabChipGX-da RNK chipini ishlatish (Kaliper hayot fanlari ).
Kichik RNKlarning gel elektroforezi bilan fraktsiyasi
Izolyatsiya qilingan RNK denatura qiluvchi poliakrilamid jelida ishlaydi. Jelning tegishli o'lchamdagi RNKni o'z ichiga olgan qismini aniqlashda, oxir-oqibat ketma-ketlikdagi material miqdorini kamaytirishda radioaktiv 5'-32P yorlig'i bilan ishlangan oligonukleotidlar kabi tasvirlash usuli qo'llaniladi. Ushbu bosqich majburiy ravishda quyida keltirilgan ligatsiya va teskari transkripsiya bosqichlaridan oldin bajarilishi shart emas.[19][20]
Ligatsiya
Bog'lanish bosqichi kichik RNKlarning ikkala uchiga DNK adapterlarini qo'shadi, ular teskari transkripsiya paytida primer bog'lanish joylari vazifasini bajaradi va PCR kuchaytirish. Adenillangan bitta zanjirli DNK 3'adaptor, so'ngra 5'adaptor T4 RNK ligaz2 kabi ligatsiya qiluvchi ferment yordamida kichik RNKlarga bog'lanadi. Adapterlar, shuningdek, 5 'gidroksil guruhi bo'lgan RNKning parchalanish mahsulotlarini emas, balki 5' fosfat guruhi, xarakterli mikroRNKlari bo'lgan kichik RNKlarni ushlash uchun mo'ljallangan.[19][20]
Teskari transkripsiya va PCR kuchaytirish
Ushbu qadam kichik adapter bog'langan RNKlarni sekvensiya reaktsiyasida ishlatiladigan cDNA klonlariga aylantiradi. Ushbu bosqichni ba'zi bir shakllardan foydalangan holda amalga oshiradigan ko'plab savdo to'plamlar mavjud teskari transkriptaz. Keyin PCR cDNA sekansiyalarini ko'paytirish uchun amalga oshiriladi. Noyob nukleotid teglari bilan ishlangan primerlardan, shuningdek, birlashtirilgan kutubxona multiplekslari ketma-ketligida ID teglarini yaratish uchun foydalanish mumkin.[19][20]
Tartiblash
Haqiqiy RNK ketma-ketligi ishlatilgan platformaga qarab sezilarli darajada o'zgarib turadi. Uchta yangi avlod avlodlari ketma-ketligi[21] platformalar Pirosekvensiya ustida 454 Hayot fanlari platforma,[22] polimeraza asosidagi ketma-ket sintez Illumina (kompaniya) platforma,[23] yoki ligatsiya bilan tartiblash ustida ABI qattiq ketma-ketligi platforma.[24]
Ma'lumotlarni tahlil qilish
MiRNA-seq ma'lumotlarini tahlil qilishda markaziylik 1) ketma-ketlik o'qishidan miRNA ko'pligi darajasini olish, 2) yangi miRNKlarni kashf etish va keyin 3) differentsial ifoda etilgan miRNA va ularning 4) bog'liq mRNA genlarining maqsadlarini aniqlash qobiliyatidir.
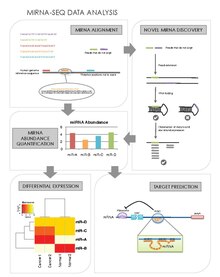
miRNA Alignment & Mouldence Quantification
miRNA'lar ba'zi hujayralar turlarida, to'qimalarida, rivojlanish bosqichlarida yoki ayniqsa saraton kabi kasallik holatlarida ifoda etilishi mumkin.[1] Chuqur sekvensiya (miRNA-seq) berilgan namunadan millionlab o'qishni hosil qilganligi sababli, bu bizga miRNKlarni profillashtirishga imkon beradi; ularning absolyut miqdorini aniqlash, ularning variantlarini kashf etish bilan bo'ladimi (ma'lum izomirlar[25]) O'qish ketma-ketligining o'rtacha uzunligi o'rtacha miRNA (17-25 nt) dan uzunroq ekanligini hisobga olsak, miRNA ning 3 'va 5' uchlari bir xil o'qishda topilishi kerak. Bir nechta miRNA miqdorini aniqlash algoritmlari mavjud.[21][26] Ularning umumiy qadamlari quyidagicha:[27]
- Sekvensiyadan so'ng xom ketma-ketlik ko'rsatkichlari sifatga qarab filtrlanadi. Adapter ketma-ketliklari ham xom ketma-ketlikni o'qishdan o'chiriladi.
- Natijada olingan o'qishlar tezkor faylga formatlanadi, bu erda har bir noyob teg uchun nusxa raqami va ketma-ketligi qayd etiladi.
- E. Coli ifloslanishini ko'rsatishi mumkin bo'lgan ketma-ketliklar a tomonidan aniqlanadi Portlash E. Coli ma'lumotlar bazasini qidirish va tahlildan olib tashlash.
- Qolgan ketma-ketliklarning har biri miRNA ketma-ketligi ma'lumotlar bazasiga (masalan, miRBase) to'g'ri keladi[28]) DICER-ning nomukammal ishlashini hisobga olish uchun 3 'uchida 6nt va 5' ning oxirida 3nt ko'tarilishga ruxsat beriladi.
- Keyin miRNA ma'lumotlar bazasiga to'g'ri kelmaydigan ko'rsatkichlar mutatsiyalarni o'tkazishi mumkin bo'lgan yoki o'tib ketgan miRNAlarni aniqlash uchun miRNA prekursorlariga yumshoq tarzda to'g'ri keladi. RNK tahriri.
- Keyin har bir miRNA uchun o'qishlar soni har bir miRNKning ko'pligi to'g'risida xabar berish uchun xaritada olingan miRNAlarning umumiy soniga normalizatsiya qilinadi.
Roman miRNA kashfiyoti
MiRNA-seqning yana bir afzalligi shundaki, u an'anaviy skrining va profil usullaridan chetda qolgan bo'lishi mumkin bo'lgan yangi miRNKlarni topishga imkon beradi.[27] Bir nechta yangi miRNA kashfiyot algoritmlari mavjud. Ularning umumiy qadamlari quyidagicha:
- Ma'lum bo'lgan miRNA ketma-ketliklariga to'g'ri kelmaydigan o'qishlarni oling va ularni genomga moslang.
- RNKni katlama usuli
- MiRNA ketma-ketliklari uchun to'liq mos keladigan narsa topildi, genomik ketma-ketlikni oling, ikkala tomonida ~ 100bp yonma-yon ketma-ketlik va RNKni Vena to'plami kabi RNK katlama dasturi orqali boshqaring.[29]
- MiRNA soch tolasining bir qo'lida joylashgan va minimal erkin energiyasi ~ 25kkal / mol dan kam bo'lgan katlamali ketma-ketliklar taxminiy miRNA sifatida ro'yxatga kiritilgan.
- Qisqa ro'yxatdagi ketma-ketliklar faqatgina mumkin bo'lgan prekursorlar ketma-ketligini o'z ichiga olgan holda qisqartiriladi va keyinchalik qo'shni ketma-ketliklar tomonidan sun'iy ravishda barqarorlashtirilmasligi uchun qayta to'ldiriladi.
- Olingan katlanmış ketma-ketliklar, agar miRNA ketma-ketligi soch tolasining bir qo'liga tushib qolsa va turlar orasida yuqori darajada saqlanib qolsa, yangi miRNA deb hisoblanadi.
- Yulduzli ipni ifodalash usuli (miRdeep[30])
- Yangi miRNA ketma-ketliklari DICERni qayta ishlash tufayli namoyon bo'ladigan xarakterli ekspression naqshlari asosida aniqlanadi: etuk miRNA ning yulduz zanjiri va pastadir ketma-ketliklari bo'yicha yuqori ifodasi.
Diferensial ifoda tahlili
Har bir namuna uchun miRNKlarning ko'pligi aniqlangandan so'ng, ularning ekspression darajalari namunalar bilan taqqoslanishi mumkin. Keyinchalik, ma'lum bir vaqt nuqtalari yoki xususan to'qimalar yoki kasallik holatlarida imtiyozli ravishda ifodalangan miRNKni aniqlash mumkin bo'ladi. Namunalar orasidagi xaritalangan o'qishlar sonini normallashtirgandan so'ng, ko'plab statistik testlardan foydalanish mumkin (ishlatilganlar kabi) gen ekspresiyasini profillash ) differentsial ifodani aniqlash uchun
Maqsadli bashorat
MiRNA ning mRNA maqsadlarini aniqlash, ular ekspressionini boshqaradigan genlar yoki genlar tarmoqlari to'g'risida tushuncha beradi.[31] Ochiq ma'lumotlar bazalari miRNA maqsadlarining prognozlarini ta'minlaydi. Ammo haqiqiy ijobiy bashoratlarni noto'g'ri ijobiy bashoratlardan yaxshiroq ajratish uchun miRNA-seq ma'lumotlarini mRNA-seq ma'lumotlariga qo'shib, miRNA: mRNA funktsional juftlarini kuzatish mumkin. RNA22,[32] TargetScan,[33][34][35][36][37][38] miRanda,[39] va PicTar[40] ushbu maqsad uchun mo'ljallangan dasturiy ta'minotdir. Bashoratli dasturlarning ro'yxati berilgan Bu yerga.Umumiy qadamlar:
- MiRNKni aniqlang: mRNA bog'lovchi juftliklar, mRNA ketma-ketligining 3'-UTR dagi miRNA sekanslari orasidagi komplementarlik aniqlanadi.
- MiRNKning saqlanish darajasini aniqlang: turlar bo'yicha mRNA bog'lovchi juftliklar. Odatda yuqori darajada bog'langan juftliklar bashorat qilishning noto'g'ri pozitsiyalari bo'lishi ehtimoli kamroq.
- MRNA-seq yoki oqsil ekspresyoni ma'lumotlarida miRNA nishonga olish dalillarini kuzatib boring: qaerda miRNA ekspresi yuqori bo'lsa, uning maqsad genining geni va oqsil ekspressioni past bo'lishi kerak.
MRNA maqsadlari uchun maqsadlarni tasdiqlash
Ko'pgina miRNAlar mRNA maqsadlarining to'g'ridan-to'g'ri parchalanishida ishlaydi; bu, ayniqsa, o'simliklarda to'g'ri keladi va shu tariqa miRNKlarning bu xususiyatidan foydalanish uchun ajratilgan yoki parchalanib ketgan mRNKlarning yopilmagan 3 'uchlarini ketma-ketlashtirish orqali yuqori rentabellikga ega bo'lgan tartiblash usullari ishlab chiqilgan. Ushbu usullar sifatida tanilgan Degradom ketma-ketligi yoki PARE.[41][42] Maxsus mRNAlarda nishon dekolatsiyasini tasdiqlash odatda 5 'ning o'zgartirilgan versiyasi yordamida amalga oshiriladi CDNA DNKlarining tez kuchayishi genga xos primer bilan.
Ilovalar
Roman miRNKlarini aniqlash
miRNA-seq ilgari an'anaviy miRNA profillash usullaridan chetga chiqqan yangi miRNKlarni aniqladi. Bunday topilmalarga misollar embrional ildiz hujayralarida,[25] tovuq embrionlari,[43] o'tkir limfoblastik leykemiya,[44] diffuz yirik b-hujayrali limfoma va b-hujayralar,[45] o'tkir miyeloid leykemiya,[46] va o'pka saratoni.[47]
Kasallik biomarkerlari
Mikro RNKlar deyarli barcha uyali jarayonlarning muhim regulyatorlari, masalan, tirik qolish, ko'payish va farqlash. Binobarin, miRNAlarning onko- va regulyatsiyasi orqali saratonning turli jihatlarida ishtirok etishi kutilmagan emas. o'smani bostiruvchi gen ifoda. Yuqori rentabellikga ega profillash usullarini ishlab chiqish bilan birgalikda miRNAlar saratonni tasniflash, terapiyaga javob va prognoz uchun biomarker sifatida aniqlandi.[48] Bundan tashqari, miRNAlar gen ekspressionini tartibga solganligi sababli, ular ma'lum bir buzuqlikni keltirib chiqarishi mumkin bo'lgan muhim tartibga soluvchi tarmoqlarda bezovtaliklarni aniqlashi mumkin.[48] Kasallikning biomarkerlari va bashoratchilari sifatida miRNAlarning bir nechta qo'llanilishi quyida keltirilgan.
| Saraton turi | miRNAlar a | Ref. |
|---|---|---|
| Ko'krak | ||
| ER holati | miR-26a / b, miR-30 oilasi, miR-29b, miR-155, miR-342, miR-206, miR-191 | [49][50][51][52] |
| PR maqomi | let-7c, miR-29b, miR-26a, miR-30 oilasi, miR-520g | [52][53] |
| HER2 /neu holat | miR-520d, miR-181c, miR-302c, miR-376b, miR-30e | [49][53] |
| O'pka | ||
| Skuamoz va skuamoz bo'lmagan hujayra | miR-205 | [54] |
| Kichik hujayra va kichik bo'lmagan hujayra | miR-17-5p, miR-22, miR-24, miR-31 | [48] |
| Oshqozon | ||
| Diffuz va ichak | miR-29b / c, miR-30 oilasi, miR-135a / b | [55] |
| Endometrium | ||
| Bachadon papilleriga qarshi endometrioid | miR-19a / b, miR-30e-5p, miR-101, miR-452, miR-382, miR-15a, miR-29c | [56] |
| Buyrak | ||
| Papillariya va hujayralarni tozalash | miR-424, miR-203, miR-31, miR-126 | [57] |
| Onkotsitoma va xromofob | miR-200c, miR-139-5p | [57] |
| Miyeloma | ||
| t bilan (14; 16) | miR-1, miR-133a | [58] |
| t (4; 14) bilan | miR-203, miR-155, miR-375 | [58] |
| t bilan (11; 14) | miR-125a, miR-650, miR-184 | [58] |
| O'tkir miyeloid leykemiya | ||
| t bilan (15; 17) | miR-382, miR-134, miR-376a, miR-127, miR-299-5p, miR-323 | [59] |
| t (8; 21) yoki inv (16) bilan | let-7b / c, miR-127 | [59] |
| bilan NPM1 mutatsiyalar | miR-10a / b, let-7, miR-29, miR-204, miR-128a, miR-196a / b | [59][60] |
| bilan FLT3 ITD | miR-155 | [59][60][61] |
| Surunkali limfotsitik leykemiya | ||
| ZAP-70 darajasi va IgVH holati | miR-15a, miR-195, miR-221, miR-155, miR-23b | [62] |
| Melanoma | ||
| BRAF V600E bilan | miR-193a, miR-338, miR-565 | [63] |
| Lenfoma | ||
| Diffuz katta B hujayrali limfoma | bor-miR-128, bor-miR-129-3p, bor-miR-152, bor-miR-155, bor-miR-185, bor-miR-193a-5p, bor-miR-196b, bor-miR- 199b-3p, has-miR-20b, has-miR-23a, has-miR-27a, has-miR-28-5p, has-miR-301a, has-miR-331-3p, has-miR-365, miR-625 ga ega, miR-9 ga ega | [45] |
aBu ushbu xavfli kasalliklarga chalingan miRNAlarning to'liq ro'yxati emas.
MiRNA profilining boshqa usullari bilan taqqoslash
MiRNA-seq-ni miRNA profilining boshqa usullariga nisbatan zararli tomonlari shundaki, u qimmatroq, odatda umumiy RNKning ko'proq miqdorini talab qiladi, keng amplifikatsiyani o'z ichiga oladi va mikroarray va qPCR usullariga qaraganda ancha vaqt talab etadi.[3] Shuningdek, miRNA-seq kutubxonasini tayyorlash usullari miRNA komplementining muntazam ravishda imtiyozli ko'rinishiga ega bo'lib tuyuladi va bu miRNA ko'pligini aniq aniqlashga to'sqinlik qiladi.[64] Shu bilan birga, yondashuv mustaqil hibridizatsiya hisoblanadi va shuning uchun uni talab qilmaydi apriori ketma-ketlik haqida ma'lumot. Shu sababli, yangi miRNA va miRNA izoformalari (izoMirs) ketma-ketligini olish, ketma-ket o'xshash miRNKlarni ajratish va nuqta mutatsiyalarini aniqlash mumkin.[65]
MiRNA profilining platformadagi taqqoslanishi
| qPCR | Mikroarray | Tartiblash | |
|---|---|---|---|
| Ishlash vaqti | ~ 6 soat | ~ 2 kun | 1-2 hafta |
| Jami RNK talab qilinadi | 500 ng | 100-1000 ng | 500-5000 ng |
| Dinamik diapazon aniqlandi | Oltita kattalik buyrug'i | To'rt daraja | Besh yoki undan ortiq buyurtma kattaligi |
| Infratuzilma va texnik talablar | Kam | O'rtacha | Muhim |
| Namuna narxi (USD) | $400 | $250–$350 | $500–$700 |
Adabiyotlar
- ^ a b Farazi, Taliya A; Spitser, Jessika I; Morozov, Pavel; Tushl, Tomas (2011). "odam saratonida miRNAlar". Patologiya jurnali. 223 (2): 102–115. doi:10.1002 / yo'l.2806. ISSN 0022-3417. PMC 3069496. PMID 21125669.
- ^ Sandhu, S .; Garzon, R. (2011). "MikroRNKlarning saraton diagnostikasi, prognozi va davolashda potentsial qo'llanilishi". Semin Onkol. 38 (6): 781–787. doi:10.1053 / j.seminoncol.2011.08.007. PMID 22082764.
- ^ a b v Beyker, Monya (2010). "MicroRNA profillash: signalni shovqindan ajratish". Tabiat usullari. 7 (9): 687–692. doi:10.1038 / nmeth0910-687. ISSN 1548-7091. PMID 20805796. S2CID 6853222.
- ^ Kim, V. Narri; Xan, Jinju; Siomi, Mikiko C. (2009). "Hayvonlardagi kichik RNKlarning biogenezi". Molekulyar hujayra biologiyasi. 10 (2): 126–139. doi:10.1038 / nrm2632. ISSN 1471-0072. PMID 19165215. S2CID 8360619.
- ^ Bartel, D (2004). "MicroRNAsGenomics, Biogenesis, Mexanizm va funktsiya". Hujayra. 116 (2): 281–297. doi:10.1016 / S0092-8674 (04) 00045-5. ISSN 0092-8674. PMID 14744438. S2CID 2669459.
- ^ a b v U, Lin; Hannon, Gregori J. (2004). "MicroRNAs: genlarni boshqarishda katta rol o'ynaydigan kichik RNKlar". Genetika haqidagi sharhlar. 5 (7): 522–531. doi:10.1038 / nrg1379. ISSN 1471-0056. PMID 15211354. S2CID 86602746.
- ^ Ambros, Viktor (1989). "Regulyativ genlar ierarxiyasi C. elegans-da lichinkadan kattalarga rivojlanish o'zgarishini boshqaradi". Hujayra. 57 (1): 49–57. doi:10.1016/0092-8674(89)90171-2. ISSN 0092-8674. PMID 2702689. S2CID 13103224.
- ^ Li, Rozalind S.; Faynbaum, Rhonda L.; Ambros, Viktor (1993). "C. elegans heterochronic gen-4 geni 14-ga antisens komplementarligi bilan kichik RNKlarni kodlaydi". Hujayra. 75 (5): 843–854. doi:10.1016 / 0092-8674 (93) 90529-Y. ISSN 0092-8674. PMID 8252621.
- ^ Uaytmen, Bryus; Xa, Ilho; Ruvkun, Gari (1993). "Lin-4 tomonidan geteroxronik genning posttranskripsiyaviy regulyatsiyasi C. elegans-da vaqtinchalik shakllanishni vositachilik qiladi".. Hujayra. 75 (5): 855–862. doi:10.1016/0092-8674(93)90530-4. ISSN 0092-8674. PMID 8252622.
- ^ Chen, C.-Z. (2004). "MicroRNAs gemotopoetik naslning farqlanishini modulyatsiya qiladi" (PDF). Ilm-fan. 303 (5654): 83–86. Bibcode:2004Sci ... 303 ... 83C. doi:10.1126 / science.1091903. hdl:1721.1/7483. ISSN 0036-8075. PMID 14657504. S2CID 7044929.
- ^ Xu, Peizhang; Vernoy, Stefani Y.; Guo, Ming; Hay, Bryus A. (2003). "Drosophila MicroRNA Mir-14 hujayra o'limini bostiradi va normal yog 'almashinuvi uchun zarur" (PDF). Hozirgi biologiya. 13 (9): 790–795. doi:10.1016 / S0960-9822 (03) 00250-1. ISSN 0960-9822. PMID 12725740. S2CID 6391484.
- ^ Johnston, Robert J.; Hobert, Oliver (2003). "Caenorhabditis elegansida chap / o'ng neyronal assimetriyani boshqaruvchi mikroRNK". Tabiat. 426 (6968): 845–849. Bibcode:2003 yil natur.426..845J. doi:10.1038 / tabiat02255. ISSN 0028-0836. PMID 14685240. S2CID 4410288.
- ^ Lee, R. C. (2001). "Caenorhabditis elegansidagi kichik RNKlarning keng klassi". Ilm-fan. 294 (5543): 862–864. Bibcode:2001 yil ... 294..862L. doi:10.1126 / science.1065329. ISSN 0036-8075. PMID 11679672. S2CID 33480585.
- ^ Aldrij, Sara; Hadfild, Jeyms (2012). MiRNA profillash texnologiyalari va platformalararo taqqoslash bilan tanishish. Molekulyar biologiya usullari. 822. Keyingi avlod MicroRNA ifodasini profillashtirish texnologiyasi. 19-31 betlar. doi:10.1007/978-1-61779-427-8_2. ISBN 978-1-61779-426-1. ISSN 1064-3745. PMID 22144189.
- ^ Lu, C; Tej, SS; Luo, S; Haudenschild, CD; Meyers, miloddan avvalgi; Green, PJ (2005 yil 2-sentabr). "Transkriptomning kichik RNK komponentini yoritib berish". Ilm-fan. 309 (5740): 1567–9. Bibcode:2005 yil ... 309.1567L. doi:10.1126 / science.1114112. PMID 16141074. S2CID 1651848.
- ^ Rubi, J. Grem; Jan, Kalvin; Aktyor, Kristofer; Axtell, Maykl J.; Li, Uilyam; Nusbaum, Chad; Ge, Hui; Bartel, Devid P. (2006). "Keng ko'lamli ketma-ketlik 21U-RNK va C. elegansdagi qo'shimcha mikroRNKlar va endogen siRNKalarni ochib beradi". Hujayra. 127 (6): 1193–1207. doi:10.1016 / j.cell.2006.10.040. ISSN 0092-8674. PMID 17174894. S2CID 16838469.
- ^ Vitten, Daniela; Tibshirani, Robert; Gu, Sem; Yong'in, Endryu; Lui, Veng-Onn (2010). "Bachadon bo'yni o'smalari va mos keladigan boshqaruv elementlari to'plamida ultra yuqori o'tkazuvchanlikni ketma-ketligiga asoslangan kichik RNK kashfiyoti va alohida statistik biomarker tahlillari". BMC biologiyasi. 8 (1): 58. doi:10.1186/1741-7007-8-58. ISSN 1741-7007. PMC 2880020. PMID 20459774.
- ^ Vu, Qian; Lu, Zuhong; Li, salomlashish; Lu, Jiafeng; Guo, Li; Ge, Qinyu (2011). "Ko'krak bezi saratonini aniqlash uchun MicroRNA-larning navbatdagi navbati". Biomeditsina va biotexnologiya jurnali. 2011: 1–7. doi:10.1155/2011/597145. ISSN 1110-7243. PMC 3118289. PMID 21716661.
- ^ a b v d Lu, C; Meyers, miloddan avvalgi; Green, PJ (oktyabr 2007). "Chuqur sekvensiya uchun kichik RNK cDNA kutubxonalarini qurish". Usullari. 43 (2): 110–7. doi:10.1016 / j.ymeth.2007.05.002. PMID 17889797.
- ^ a b v d e Xafner, Markus; Landgraf, Pablo; Lyudvig, Yanos; Rays, Amanda; Ojo, Tolulope; Lin, Karolina; Xoloch, Doniyor; Lim, Sindi; Tushl, Tomas (2008). "CDNA kutubxonasi ketma-ketligi yordamida mikroRNKlarni va boshqa kichik tartibga soluvchi RNKlarni aniqlash". Usullari. 44 (1): 3–12. doi:10.1016 / j.ymeth.2007.09.009. ISSN 1046-2023. PMC 2847350. PMID 18158127.
- ^ a b Shendure, Jey; Dji, Xanli (2008). "Keyingi avlod DNK sekvensiyasi". Tabiat biotexnologiyasi. 26 (10): 1135–1145. doi:10.1038 / nbt1486. ISSN 1087-0156. PMID 18846087. S2CID 6384349.
- ^ "Arxivlangan nusxa". Arxivlandi asl nusxasi 2011-05-26. Olingan 2012-03-01.CS1 maint: nom sifatida arxivlangan nusxa (havola)
- ^ http://www.illumina.com/pages.ilmn?ID=204
- ^ "Arxivlangan nusxa". Arxivlandi asl nusxasi 2008-05-16. Olingan 2008-05-16.CS1 maint: nom sifatida arxivlangan nusxa (havola)
- ^ a b Morin, R. D .; O'Konnor, M. D .; Griffit, M.; Kuchenbauer, F.; Delani, A .; Prabxu, A.-L .; Chjao, Y .; Makdonald, X.; Zeng, T .; Xirst, M.; Eaves, C. J .; Marra, M. A. (2008). "Insonning embrional ildiz hujayralarida mikroRNK profillashi va kashfiyotiga massiv parallel ketma-ketlikni qo'llash". Genom tadqiqotlari. 18 (4): 610–621. doi:10.1101 / gr.7179508. ISSN 1088-9051. PMC 2279248. PMID 18285502.
- ^ Berninger, Filipp; Gaidatzis, Dimos; van Nimvegen, Erik; Zavolan, Mixaela (2008). "Kichik RNK klonlash ma'lumotlarini hisoblash tahlili". Usullari. 44 (1): 13–21. doi:10.1016 / j.ymeth.2007.10.002. ISSN 1046-2023. PMID 18158128.
- ^ a b Kreyton, C. J .; Reid, J. G.; Gunaratne, P. H. (2009). "MikroRNKlarni chuqur sekvensiya yordamida ifodalash profilingi". Bioinformatika bo'yicha brifinglar. 10 (5): 490–497. doi:10.1093 / bib / bbp019. ISSN 1467-5463. PMC 2733187. PMID 19332473.
- ^ Kozomara, A .; Griffits-Jons, S. (2010). "miRBase: microRNA annotatsiyasi va chuqur ketma-ketlik ma'lumotlarini birlashtirish". Nuklein kislotalarni tadqiq qilish. 39 (Ma'lumotlar bazasi): D152-D157. doi:10.1093 / nar / gkq1027. ISSN 0305-1048. PMC 3013655. PMID 21037258.
- ^ Xofaker, I. L .; Fontana, V.; Stadler, P. F.; Bonxeffer, L. S .; Taker, M.; Schuster, P. (1994). "RNK ikkilamchi tuzilmalarini tez katlama va taqqoslash". Monatshefte für Chemie. 125 (2): 167–188. doi:10.1007 / BF00818163. ISSN 0026-9247. S2CID 19344304.
- ^ Yang X.; Li, L. (2011). "miRDeep-P: o'simliklarda mikroRNA transkriptomini tahlil qilish uchun hisoblash vositasi". Bioinformatika. 27 (18): 2614–5. doi:10.1093 / bioinformatika / btr430. ISSN 1367-4803. PMID 21775303.
- ^ Klunan, Nikol; Vani, Shivangi; Xu, Qinying; Gu, Tszian; Lea, Kristi; Isitgich, Sheila; Barbacioru, Katalin; Stepto, Anita L; Martin, Xilari S; Nurbaxsh, Ehsan; Krishnan, Keertana; Gardiner, Bruk; Vang, Xiaohui; Nons, Katiya; Stin, Jeyson A; Matigan, Nik; Vud, Devid L; Kassaxn, Karin S; Vaddell, Nic; Cho'pon, Jil; Li, Klarens; Ichikava, Jef; MakKernan, Kevin; Bramlett, Kelli; Kuersten, Skott; Grimmond, Shon M (2011). "MicroRNAs va ularning izomiRlari umumiy biologik yo'llarni nishonga olish uchun birgalikda ishlaydi". Genom biologiyasi. 12 (12): R126. doi:10.1186 / gb-2011-12-12-r126. ISSN 1465-6906. PMC 3334621. PMID 22208850.
- ^ Miranda KC, Huynh T, Tay Y, Ang YS, Tam WL, Tomson AM, Lim B, Rigoutsos I (2006). "MicroRNA ulanish joylari va ularga mos keladigan heteroduplekslarni aniqlash uchun naqshga asoslangan usul". Hujayra. 126 (6): 1203–17. doi:10.1016 / j.cell.2006.07.031. PMID 16990141. S2CID 12749133.
- ^ Lyuis, BP; Burge CB; Bartel DP (2005 yil 14-yanvar). "Odatda adenozinlar bilan yonma-yon turadigan urug'larni bir-biriga bog'lab qo'yish, odamlarning minglab genlari mikroRNK nishonlari ekanligidan dalolat beradi". Hujayra. 120 (1): 15–20. doi:10.1016 / j.cell.2004.12.035. PMID 15652477. S2CID 17316349.
- ^ Grimson, A; Farx, KK; Johnston, WK; Garret-Engele, P; Lim, LP; Bartel, DP (6 Jul, 2007). "Sutemizuvchilarning o'ziga xos xususiyatiga ega bo'lgan MicroRNA: urug'larni juftlashtirishdan tashqari determinantlar". Molekulyar hujayra. 27 (1): 91–105. doi:10.1016 / j.molcel.2007.06.017. PMC 3800283. PMID 17612493.
- ^ Fridman, RC; Farx, KK; Burge, CB; Bartel, DP (yanvar, 2009 yil). "Ko'pgina sutemizuvchilar mRNKlari mikroRNKlarning saqlanib qolgan maqsadlari". Genom tadqiqotlari. 19 (1): 92–105. doi:10.1101 / gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Garsiya, DM; Baek, D; Shin, C; Bell, GW; Grimson, A; Bartel, DP (2011 yil 11 sentyabr). "Urug'larni juftlashtirishning zaif barqarorligi va mo'ljaldagi mo'l-ko'llik lsy-6 va boshqa mikroRNKlarning malakasini pasaytiradi". Tabiatning strukturaviy va molekulyar biologiyasi. 18 (10): 1139–46. doi:10.1038 / nsmb.2115. PMC 3190056. PMID 21909094.
- ^ Agarval, Vikram; Bell, Jorj V.; Nam, Jin-Vu; Bartel, Devid P. (2015-08-12). "Sutemizuvchilar mRNKlarida samarali mikroRNK nishon joylarini bashorat qilish". eLife. 4: e05005. doi:10.7554 / eLife.05005. ISSN 2050-084X. PMC 4532895. PMID 26267216.
- ^ Agarval, V; Subtelny, AO; Thiru, P; Ulitskiy, men; Bartel, DP (4 oktyabr 2018). "Drozofilada mikroRNKning samaradorligini taxmin qilish". Genom biologiyasi. 19 (1): 152. doi:10.1186 / s13059-018-1504-3. PMC 6172730. PMID 30286781.
- ^ Mazier, P; Enright, A (2007). "MikroRNK maqsadlarini bashorat qilish". Bugungi kunda giyohvand moddalarni kashf etish. 12 (11–12): 452–458. doi:10.1016 / j.drudis.2007.04.002. ISSN 1359-6446. PMID 17532529.
- ^ Krek, A. MikroRNK maqsadlarini aniqlash. DAI-B 70/07, (2010).
- ^ Nemis MA, Pillay M, Jeong DH, Hetawal A, Luo S, Janardhanan P, Kannan V, Rymarquis LA, Nobuta K, German R, De Paoli E, Lu C, Schroth G, Meyers BC, Green PJ (2008). "RNK uchlarini parallel tahlil qilish orqali mikroRNK-maqsadli RNK juftlarini global identifikatsiyasi". Nat. Biotexnol. 26 (8): 941–946. doi:10.1038 / nbt1417. PMID 18542052. S2CID 13187064.
- ^ Addo-Quaye C, Eshoo TW, Bartel DP, Axtell MJ (2008). "Arabidopsis degradomining ketma-ketligi bilan aniqlangan endogen siRNA va miRNA maqsadlari". Curr. Biol. 18 (10): 758–762. doi:10.1016 / j.cub.2008.04.042. PMC 2583427. PMID 18472421.
- ^ Buermans, Xenk PJ; Ariyurek, Yavuz; van Ommen, Gertjan; den Dunnen, Yoxan T; Hoen, Piter AC (2010). "Keyingi avlod ketma-ketligini mikroRNK ekspresiyasini profillashtirishning yangi usullari". BMC Genomics. 11 (1): 716. doi:10.1186/1471-2164-11-716. ISSN 1471-2164. PMC 3022920. PMID 21171994.
- ^ Chjan, Baoxon; Chjan, Xua; Yang, Tszian-Xua; Chjen, Yu-Sheng; Chjan, Peng; Chen, Xiao; Vu, iyun; Xu, Ling; Luo, Xue-Tsun; Ke, Chji-Yong; Chjou, Xuy; Qu, Liang-Xu; Chen, Yue-Qin (2009). "Keng sekvensiya yondashuvi asosida insonning o'tkir limfoblastik leykemiyasida kichik RNK va yangi mikro-RNK kashfiyotlarining genom-keng tahlili". PLOS ONE. 4 (9): e6849. Bibcode:2009PLoSO ... 4.6849Z. doi:10.1371 / journal.pone.0006849. ISSN 1932-6203. PMC 2731166. PMID 19724645.
- ^ a b Jima, D. D.; Chjan, J .; Jeykobs, C .; Richards, K. L .; Dunfi, C. X.; Choi, V. V. L.; Yan Au, V.; Srivastava, G.; Czader, M. B .; Rizzieri, D. A .; Lagoo, A. S .; Lugar, P. L .; Mann, K. P.; Gullar, C. R .; Bernal-Mizrachi, L.; Naresh, K. N .; Evens, A. M .; Gordon, L. I .; Luftig, M .; Fridman, D. R .; Vaynberg, J. B .; Tompson, M. A .; Gill, J. I .; Liu, Q .; Qanday qilib, T .; Grubor, V .; Gao, Y .; Patel, A .; Vu, H.; Chju, J .; Blobe, G. C .; Lipskiy, P. E.; Chadbern, A .; Deyv, S. S. (2010). "Oddiy va zararli B hujayralarining kichik RNK transkriptomini chuqur ketma-ketligi yuzlab yangi mikroRNKlarni aniqlaydi". Qon. 116 (23): e118-e127. doi:10.1182 / qon-2010-05-285403. ISSN 0006-4971. PMC 3012600. PMID 20733160.
- ^ Starchinowski, D. T.; Morin, R .; McPherson, A .; Lam, J .; Chari, R .; Wegrzyn, J .; Kuchenbauer, F.; Xirst, M.; Toxima, K .; Hamfris, R. K .; Lam, V. L.; Marra, M.; Karsan, A. (2010). "Leykemiya bilan bog'liq genomik o'zgarishlarda joylashgan odam mikroRNKlarini genom bo'yicha aniqlash". Qon. 117 (2): 595–607. doi:10.1182 / qon-2010-03-277012. ISSN 0006-4971. PMID 20962326.
- ^ Keller, Andreas; Backes, Kristina; Leydinger, Petra; Kefer, Natali; Boysgerin, Valeska; Barbacioru, Katalin; Vogel, Britta; Matzas, Mark; Xuver, Xanno; Katus, Gyugo A.; Styler, Kord; Meder, Benjamin; Meese, Ekart (2011). "Keyingi avlod ketma-ketligi o'pka saratoni bilan kasallangan bemorlarning periferik qonidagi yangi mikroRNKlarni aniqlaydi". Molekulyar biosistemalar. 7 (12): 3187–99. doi:10.1039 / c1mb05353a. ISSN 1742-206X. PMID 22027949.
- ^ a b v Chan, Elsi; Prado, Daniel Estéves; Weidhaas, Joanne Barnes (2011). "Saraton mikroRNKlari: pastki turini profillashdan terapiya reaktsiyasini bashorat qilishgacha". Molekulyar tibbiyot tendentsiyalari. 17 (5): 235–243. doi:10.1016 / j.molmed.2011.01.008. ISSN 1471-4914. PMC 3092835. PMID 21354374.
- ^ a b Blenkiron, Cheri; Goldstein, Leonard D; Torn, Natali P; Spiteri, Inmakulada; Chin, Suet-Feun; Dunning, Mark J; Barbosa-Morais, Nuno L; Teschendorff, Endryu E; Yashil, Endryu R; Ellis, Yan O; Tavaré, Simon; Kaldas, Karlos; Miska, Erik A (2007). "Odamning ko'krak bezi saratonining mikroRNK ekspression profilingi o'smaning pastki turining yangi belgilarini aniqlaydi". Genom biologiyasi. 8 (10): R214. doi:10.1186 / gb-2007-8-10-r214. ISSN 1465-6906. PMC 2246288. PMID 17922911.
- ^ Sempere, L. F .; Kristensen, M .; Silaxatoglu, A .; Bak, M.; Xit, C. V .; Shvarts, G.; Uells, V.; Kauppinen, S .; Cole, C. N. (2007). "Ko'krak bezi saratonida epiteliya hujayralarining o'ziga xos subpopulyatsiyalari bilan chegaralangan mikroRNKning o'zgarishi". Saraton kasalligini o'rganish. 67 (24): 11612–11620. doi:10.1158 / 0008-5472. CAN-07-5019. ISSN 0008-5472. PMID 18089790.
- ^ Matti, Maykl D; Benz, Kristofer S; Bowers, Jessica; Sensinger, Kelli; Vong, Linda; Skott, Gari K; Fedele, Vita; Ginzinger, Devid; Gets, Robert; Haqq, Kris (2006). "Optimallashtirilgan yuqori o'tkazuvchan mikroRNK ekspresiyasi profilaktikasi prostata va ko'krak bezi saratoni biopsiyalarining yangi biomarker bahosini beradi". Molekulyar saraton. 5 (1): 24. doi:10.1186/1476-4598-5-24. ISSN 1476-4598. PMC 1563474. PMID 16784538.
- ^ a b Iorio, M. V .; Ferracin, M.; Liu, C.-G.; Veronese, A .; Spizzo, R .; Sabbioni, S .; Magri, E .; Pedriali, M .; Fabbri, M .; Kempiglio, M .; Menard, S .; Palazzo, J. P .; Rozenberg, A .; Musiani, P .; Voliniya, S .; Nensi, I .; Kalin, G. A .; Querzoli, P .; Negrini, M .; Croce, C. M. (2005). "Odamning ko'krak bezi saratonida MicroRNA gen ekspressionini tartibga solish". Saraton kasalligini o'rganish. 65 (16): 7065–7070. doi:10.1158 / 0008-5472. CAN-05-1783. ISSN 0008-5472. PMID 16103053.
- ^ a b Loweri, Aoife J; Miller, Nikola; Devani, Amanda; Makneyl, Ruzin E; Davoren, Pamela A; Lemetr, Kristof; Benes, Vladimir; Shmidt, Sabin; Bleyk, Jonaton; To'p, Grem; Kerin, Maykl J (2009). "MicroRNA imzolari ko'krak bezi saratonida estrogen retseptorlari, progesteron retseptorlari va HER2 / neu retseptorlari holatini taxmin qiladi". Ko'krak bezi saratonini o'rganish. 11 (3): R27. doi:10.1186 / bcr2257. ISSN 1465-5411. PMC 2716495. PMID 19432961.
- ^ Livan, D .; Benjamin, X.; Gilad, S .; Ezagouri, M .; Dov, A .; Ashkenazi, K .; Gefen, N .; Izraeli, S .; Rechavi, G .; Pass, H.; Nonaka, D .; Li, J .; Spektor, Y .; Rozenfeld, N .; Chajut, A .; Koen, D .; Aharonov, R .; Mansuxani, M. (2009). "Hsa-miR-205 ifodasi asosida diagnostik tahlil skuamozni noaniq kam bo'lmagan kichik hujayrali o'pka karsinomasidan ajratib turadi". Klinik onkologiya jurnali. 27 (12): 2030–2037. doi:10.1200 / JCO.2008.19.4134. ISSN 0732-183X. PMID 19273703.
- ^ Ueda, Tetsuya; Voliniya, Stefano; Okumura, Xirosi; Shimizu, Masayoshi; Tachioli, Kristian; Rossi, Simona; Alder, Xansyuerg; Lyu, Chang-gong; Oue, Naohid; Yasui, Vataru; Yoshida, Kazuxiro; Sasaki, Xiroki; Nomura, Sachiyo; Seto, Yasuyuki; Kaminishi, Michio; Kalin, Jorj A; Croce, Carlo M (2010). "MikroRNK ekspressioni va oshqozon saratoni progressiyasi va prognozi o'rtasidagi bog'liqlik: mikroRNK ekspression tahlili". Lanset onkologiyasi. 11 (2): 136–146. doi:10.1016 / S1470-2045 (09) 70343-2. ISSN 1470-2045. PMC 4299826. PMID 20022810.
- ^ Ratner, Elena S.; Tuck, David; Rixter, Kristin; Nallur, Sunita; Patel, Rajeshvari M.; Shultz, Vins; Xui, Pei; Shvarts, Piter E.; Rezerford, Tomas J.; Weidhaas, Joanne B. (2010). "MicroRNA imzolari bachadon saratoni o'simtasining pastki turlarini ajratib turadi". Ginekologik onkologiya. 118 (3): 251–257. doi:10.1016 / j.ygyno.2010.05.010. ISSN 0090-8258. PMC 2918705. PMID 20542546.
- ^ a b Fridman, Eddi; Dotan, Zoxar; Barshak, Iris; Devid, Miriyam Ben; Dov, Avital; Tabak, Sarit; Sion, Orit; Benjamin, Sima; Benjamin, Xila; Kuker, Xagit; Avivi, Kamila; Rozenblatt, Kinneret; Polak-Charcon, Silvi; Ramon, Yoqub; Rozenfeld, Nitsan; Spector, Yael (2010). "MicroRNA ekspressioni yordamida buyrak o'smalarining aniq molekulyar tasnifi". Molekulyar diagnostika jurnali. 12 (5): 687–696. doi:10.2353 / jmoldx.2010.090187. ISSN 1525-1578. PMC 2928434. PMID 20595629.
- ^ a b v Gutierrez, N C; Sarasquete, M E; Misevich-Krzeminska, men; Delgado, M; De Las Rivas, J; Ticona, F V; Ferminan, E; Martin-Ximenes, P; Chillon, C; Risueño, A; Ernandes, J M; Garsiya-Sanz, R; Gonsales, M; San-Migel, J F (2010). "Multipl miyelomning turli xil genetik subtiplarida mikroRNK ekspresiyasining regulyatsiyasi va genlarning ekspression profilatsiyasi bilan o'zaro bog'liqlik". Leykemiya. 24 (3): 629–637. doi:10.1038 / leu.2009.274. ISSN 0887-6924. PMID 20054351.
- ^ a b v d Yongen-Lavrencich, M.; Quyosh, S. M .; Dijkstra, M. K .; Valk, P. J. M.; Lowenberg, B. (2008). "O'tkir miyeloid leykemiyaning genetik heterojenligi bilan bog'liq ravishda mikro-RNK ekspressioni". Qon. 111 (10): 5078–5085. doi:10.1182 / qon-2008-01-133355. ISSN 0006-4971. PMID 18337557.
- ^ a b Garzon, R .; Garofalo, M .; Martelli, M. P.; Brizevits, R .; Vang, L .; Fernandez-Saymering, S.; Voliniya, S .; Liu, C.-G.; Shnittger, S .; Xaferlax, T .; Liso, A .; Diverio, D.; Manchini, M .; Meloni G.; Foa, R .; Martelli, M. F.; Mecucci, C .; Croce, C. M .; Falini, B. (2008). "Sitoplazmatik mutatsiyaga uchragan nukleofosminni o'z ichiga olgan o'tkir miyeloid leykemiyaning o'ziga xos mikroRNK imzosi". Milliy fanlar akademiyasi materiallari. 105 (10): 3945–3950. Bibcode:2008 yil PNAS..105.3945G. doi:10.1073 / pnas.0800135105. ISSN 0027-8424. PMC 2268779. PMID 18308931.
- ^ Marcucci, Gido; Radmaxer, Maykl D. Maharri, Kati; Mrozek, Kshishtof; Ruppert, Emi S.; Paschka, Piter; Vukosavlyevich, Tamara; Uitman, Syuzan P.; Baldus, Klaudiya D.; Langer, nasroniy; Lyu, Chang-Gong; Kerol, Endryu J.; Pauell, Bayard L.; Garzon, Ramiro; Kros, Karlo M.; Kolits, Jonathan E.; Kaligiuri, Maykl A .; Larson, Richard A.; Bloomfild, Klara D. (2008). "Sitogenetik jihatdan normal o'tkir Miyeloid leykemiyada mikroRNK ifodasi". Nyu-England tibbiyot jurnali. 358 (18): 1919–1928. doi:10.1056 / NEJMoa074256. ISSN 0028-4793. PMID 18450603.
- ^ Kalin, Jorj Adrian; Ferracin, Manuela; Cimmino, Ameliya; Di Leva, Gianpiero; Shimizu, Masayoshi; Voychik, Silviya E.; Iorio, Marilena V.; Visone, Roza; Sever, Nurettin Ilfer; Fabbri, Myuller; Juliano, Rodolfo; Palumbo, Tiziana; Pichiorri, Flaviya; Roldo, Klaudiya; Garzon, Ramiro; Sevignani, Cinzia; Rassenti, Laura; Alder, Xansyuerg; Voliniya, Stefano; Lyu, Chang-gong; Kipps, Tomas J.; Negrini, Massimo; Croce, Carlo M. (2005). "Surunkali limfotsitik leykemiyada prognoz va progressiya bilan bog'liq bo'lgan MicroRNA imzosi". Nyu-England tibbiyot jurnali. 353 (17): 1793–1801. doi:10.1056 / NEJMoa050995. ISSN 0028-4793. PMID 16251535.
- ^ Karamuta, Stefano; Egyhazi, Suzanna; Rodolfo, Monika; Vitten, Daniela; Xansson, Yoxan; Larsson, Katarina; Lui, Veng-Onn (2010). "Xavfli melanomada mutatsion holat va omon qolish bilan bog'liq bo'lgan MicroRNA ifodalari". Tergov dermatologiyasi jurnali. 130 (8): 2062–2070. doi:10.1038 / jid.2010.63. ISSN 0022-202X. PMID 20357817.
- ^ Linsen, Sem E V; de Wit, Elzo; Yanssen, Jorj; Isitgich, Sheila; Chapman, Laura; Parkin, Rachael K; Fritz, Brayan; Vayman, Stacia K; de Bryuyn, Evart; Voest, Emil E; Kuersten, Skott; Tewari, Muneesh; Kuppen, Edvin (2009). "Kichik RNK raqamli gen ekspression profilining cheklashlari va imkoniyatlari". Tabiat usullari. 6 (7): 474–476. doi:10.1038 / nmeth0709-474. ISSN 1548-7091. PMID 19564845. S2CID 7953265.
- ^ Git, A .; Dvinge, X.; Salmon-Divon, M.; Osborne, M .; Kutter, C .; Hadfild, J .; Bertone, P .; Caldas, C. (2010). "Mikroarray profillashni, real vaqtda PCR va differentsial mikroRNK ekspresiyasini o'lchash uchun keyingi avlod ketma-ketligini texnologiyasini muntazam taqqoslash". RNK. 16 (5): 991–1006. doi:10.1261 / rna.1947110. ISSN 1355-8382. PMC 2856892. PMID 20360395.
